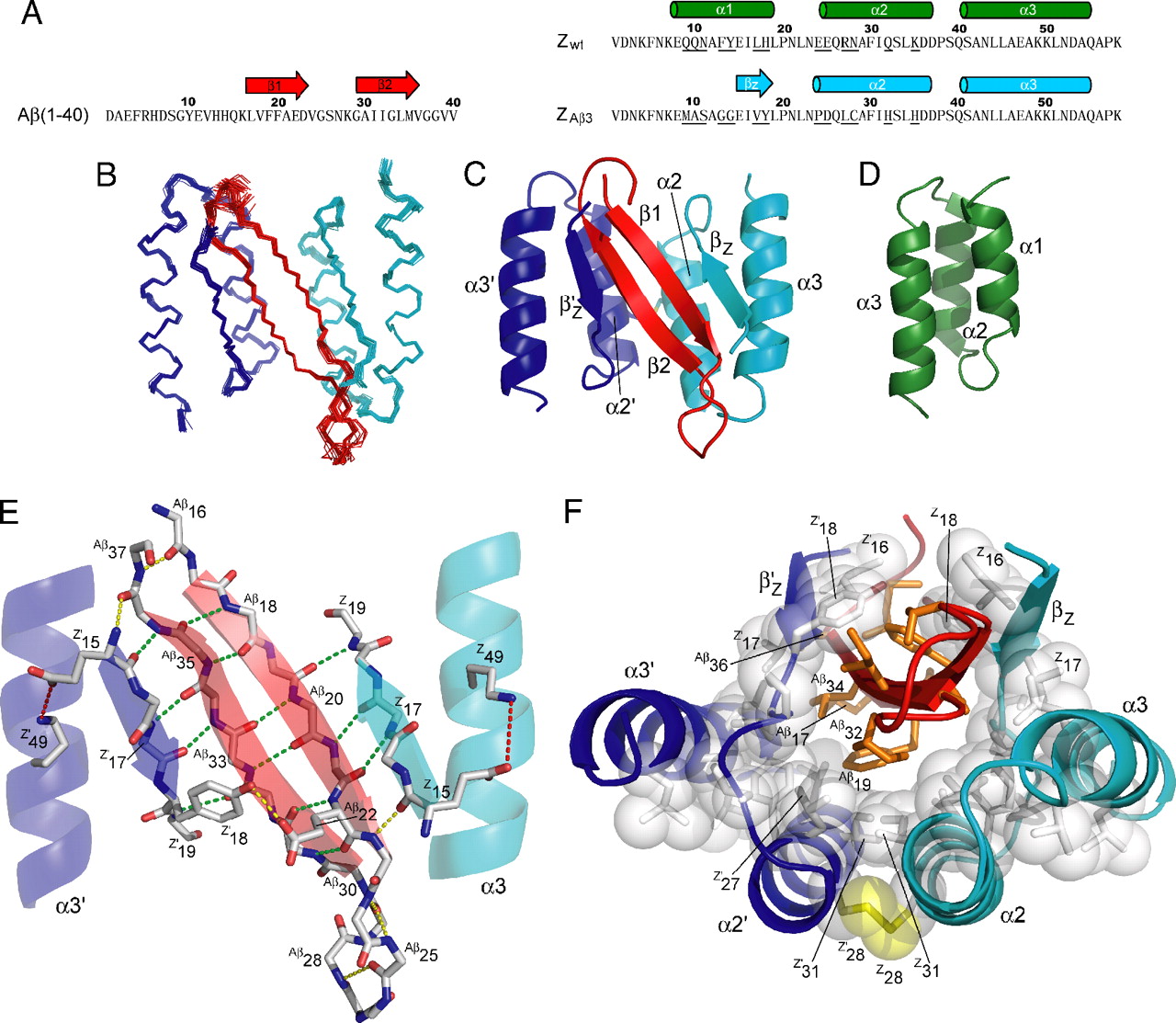
Å unnslippe tilfeldighet: Kan folding forklare proteinfunksjonelle former?
Evolutionews; 23. juni 2017
Bilde 1: Proteiner, Kreditt: Illustra Media
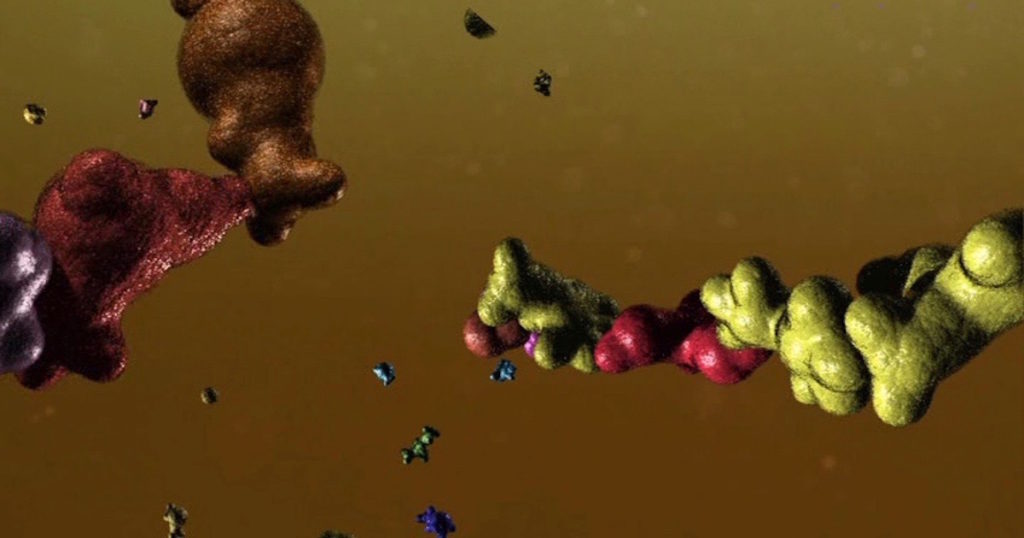
Inspirerer emnet proteinfolding deg? Les dette for å se hvorfor det kanskje burde:
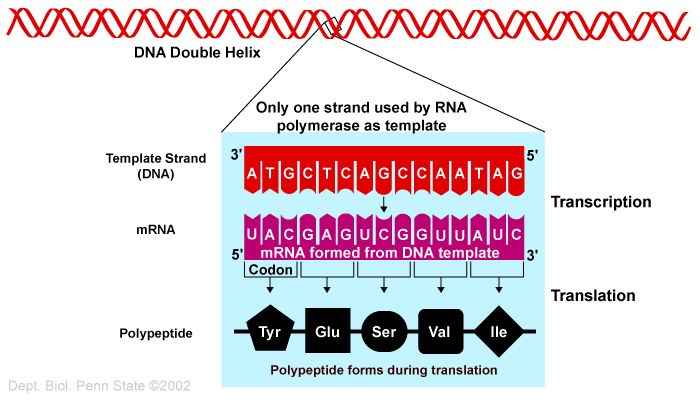
"Proteinfolding er blant de viktigste reaksjonene i all biologi . Imidlertid 50 år etter CB Anfinsen viste at proteiner kan foldes spontant uten hjelp utenfra, og til tross for det intensive arbeidet med tusenvis av forskere som fører til mer enn fem publikasjoner per dag i dagens litteratur, er det fortsatt ingen generell enighet om de mest grunnleggende spørsmålene. Hvordan folder proteiner seg? Hvorfor folder de seg på den måten? Hvordan er rekkefølgen av folding kodet i en 1D-aminosyresekvens? Disse spørsmålene har grunnleggende betydning for protein-vitenskap og dens mange anvendelser. Gjennom årene har disse spørsmålene generert en stor litteratur som fører til forskjellige modeller for foldingsprosessen."
Kort sagt, ditt liv er avhengig av proteinfolding, og emnet medbringer en klassisk konkurranse mellom intelligent design og vitenskapelig materialisme. Det er nok til å få en omtenksom person å legge merke til det.
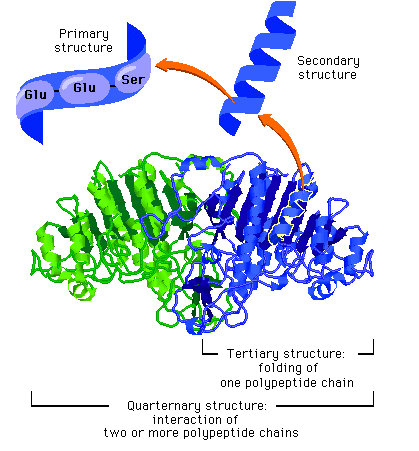
Den siterte passasjen kommer fra en artikel i Proceedings of National Academy of Science -her, av to biofysikere ved University of Pennsylvania. De så gjennom det store volumet av litteratur om emnet for å vurdere de beste nåværende modellene for å forklare hvordan én-dimensjonale sekvenser av aminosyrer kan ende opp som tredimensjonale former som utfører funksjonelt arbeid. For å sette pris på utfordringen, prøv å samle en streng perler, hvorav noen har elektriske ladninger eller tiltrekning til vann, som når det er høvelig, spontant vil 'gjøre seg om' til et verktøy. Dine celler gjør noe slikt hele tiden, og gjør det vanligvis riktig.
Biologisk institutt-forsker Douglas Axe har jobbet med problemet proteinfolding i mye av karrieren sin. Han har blitt tilsluttet av en annen forsker, Discovery Instituttets Ann Gauger, for å vise hvorfor proteinfolding gir evidens for intelligent design. Emnet blir også omtalt i Axes siste bok, Undeniable: Hvordan 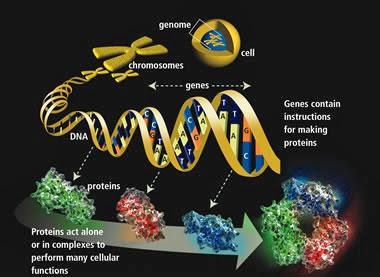 biologi bekrefter vår intuisjon som livet er utformet (Harper One, 2016) -lenke.
biologi bekrefter vår intuisjon som livet er utformet (Harper One, 2016) -lenke.
Her er problemet for materialisme i et nøtteskall: Antall måter du kan sette sammen aminosyrer som ikke foldes, overskrider stort antall måter som vil brettes. For å forvente at en tilfeldig prosess skal utforske "sekvensrommet" (settet av alle sekvenser av aminosyrer) og komme til en som folder seg, er så høyst usannsynlig at det sannsynligvis aldri vil forekomme i flere univers. Axe fulgte Michael Dentons forutanelse at "funksjonelle proteiner kan være svært sjeldne" og satte noen tall på det. Han fastsatte at det bare er "én god proteinsekvens for hver 10^74 dårlige" ( Undeniable , s. 57). Dette var om lag 10 millioner milliarder milliarder ganger mer usannsynlig enn Dentons opprinnelige estimat.
Som Axe fortsetter med å si, materialistene har ikke akkurat satt "ute av virksomhet" tegn på sine dører, da han publiserte sine resultater. Det bringer oss til nåværende artikkel - et av de siste forsøkene på å finne en måte å unngå implikasjonene av design og finne et naturlig, ikke-styrt middel for å søke sekvensrommet for de unnvikende foldene.
Bilde 2. Kun meget få kombinasjoner vil gi fungerende protein
Forfatterne, S. Walter Englander og Leland Mayne, vet altfor godt at tilfeldige søk er håpløst. Selv på 1990-tallet hadde "Levinthal bidratt til den endelige observasjonen at et tilfeldig søk ikke kunne gjøre rede for kjente foldingsrater." De fleste proteiner finner sin 'medfødte' (native) fold ekstremt raskt - noen i løpet av mikrosekunder. Noen trenger litt hjelp fra en "anstand" som GRO-EL som tillater at polypeptidet foldes i et tønne-formet kammer. I begge tilfeller vet forfatterne at tilfeldige forsøk på å finne den riktige eller "opprinnelige" -folden, selv for et korrekt sekvensert polypeptid, ville være altfor sakte hvis det var mange veier til riktig fold. Dette førte forskere tidlig til å mistenke at proteiner følger et energilandskap som knytter dem til den medfødte fold, akkurat som en trakt fører baller ned til et smalt hull. Ballen kan hoppe rundt i trakten, men formen på energilandskapet tvinger den i riktig retning. Dette er kjent som Energi Landskap Teori (ELT).
"Et kritisk trekk ved trakte-ELT-modellen er at konvensjonelle søk på mange veier på restnivå må være 'trukket mot' natur-liknende interaksjoner. Ellers, som nevnt av Levinthal (57), ville et uønsket tilfeldig søk kreve svært lang tid. Hvordan denne påvirkning kan bli implementert i form av ekte proteininteraksjoner, har aldri blitt oppdaget."
Forfatterne er ikke fornøyd med evolusjonære 'det bare er slik' historier:
"Man hevder ganske enkelt at naturlig evolusjon har gjort det slik, formulerer denne oppfatningen som et såkalt 'prinsipp om minimal frustrasjon', og tilskriver den til formen på det trakformede energi-landskapet. Proteiner "vet" på et eller annen ukjent vis hvordan de skal gjøre riktige valg."
Beklager, ingen terninger.
"En beregning av Zwanzig et al. på det aller viktigste nivået, kvantifiserer energi-avviket som ville være nødvendig. For at proteiner skal foldes i en rimelig tidsskala, må den frie energi-avviket mot korrekt i motsetning til ukorrekte vekselvirkninger, uansett hvor foldene er, være 2 kT (1,2 kcal/mol). Den entalpiske (enthalpic) forstyrrelse mellom korrekte og feilaktige interaksjoner må være enda større, godt over 2 kcal/mol, fordi konkurranse med det store entropiske havet av feil-alternativer er så ugunstig. Kjente aminosyre interaksjons-energier, mindre enn 1 kcal/mol (59), synes å gjøre denne graden av selektivitet umulig på rest-restnivået."
Er vi inspirerte enda? Dette blir veldig interessant. Spenningen vokser. Med tilfeldighet ut av spørsmålet, hva vil de gjøre?
De tar i utgangspunktet en 'splitt-og-hersk' tilnærming. Å få et stort polypeptid til å foldes seg, er for vanskelig, men kanskje hvis de kan bryte problemet ned i bit-størrelser, kan de komme til målet uten intelligens. Tross alt er det mye lettere å strikke en genser dersom firkant-mønstrene kommer ferdige, slik at du ikke trenger å lage dem fra bunnen av. "Kvantisert" på denne måten, blir problemet mer håndterlig.
"De strukturelle enhetene som samler kinetiske mellomprodukter er stort sett de samme som de kooperative bygnings-blokkene til det opprinnelige proteinet. Denne strategien deler det kinetiske puslespillet i en sekvens av mindre puslespill, danner stykker av den opprinnelige strukturen og setter dem på plass i en trinnvis vei (figur 1B). Dette er den definerte gangvei-modellen."
Bilde 3. Polypeptid under utforming
 De gir navnet "foldon" til en liten kjede av aminosyrer "kanskje 15 til 35 i antall" som folder et lite stykke. Hvis polypeptidet er sammensatt av et antall av disse prefabrikerte foldon's, vil hele proteinet kanskje finne sin opprinnelige fold raskt og synke i trakten på en trinnvis måte. Eksperimenter som utfolder og refolder noen proteiner, viser faktisk denne typen trinnvis energilandskap. De liker at:
De gir navnet "foldon" til en liten kjede av aminosyrer "kanskje 15 til 35 i antall" som folder et lite stykke. Hvis polypeptidet er sammensatt av et antall av disse prefabrikerte foldon's, vil hele proteinet kanskje finne sin opprinnelige fold raskt og synke i trakten på en trinnvis måte. Eksperimenter som utfolder og refolder noen proteiner, viser faktisk denne typen trinnvis energilandskap. De liker at:
"Hensikten med denne artikkelen er å vurdere den nåværende statusen til disse ganske forskjellige modellene og forholde dem til de sentrale spørsmålene om proteinfolding - hvordan, hvorfor og kodingsproblemet. Vi foreslår å stole på det solide grunnlaget for eksperimentet i stedet for de utallige mindre definitive forslagene og slutninger som så ofte har vært brukt i dette vanskelige feltet."
Empirisk fasthet; Hva er galt ved det? Så istedenfor å forestille seg en korrekt sekvens av aminosyrer fra grunnen av, erstatter de en sekvens av foldoner, og øker sannsynligheten for å fullføre søket i tide. Vil dette fungere i evolusjonære termer?
"Den motsatte, definerte banemodellen stammer fra eksperimentelle resultater som viser at proteiner er sammenstillinger av små kooperative enheter kalt foldoner, og at et antall proteiner folder i en reproduserbar bane, en foldenhet om gangen. Således koder de samme foldon-interaksjonene som koder for den opprinnelige strukturen av et gitt protein, også naturlig for sin spesielle foldon-baserte foldingsbane, og de virker sammen for å produsere energi-avviket mot opprinnelige interaksjoner som er nødvendige for effektiv folding."
Så hvordan, nøyaktig, dukket denne klare løsningen opp, uten intelligens?
"Tilgjengelig informasjon antyder at kvantisert opprinnelig struktur og trinnvis folding ko-evoluerte i gamle repetisjons-proteiner og ble beholdt som et funksjonelt par på grunn av deres anvendelighet for å løse det vanskelige proteinfoldingsproblemet."
"Ko-evolusjon" igjen. Så mye for empirisk strikthet. De er tilbake til 'det bare er slik' fortellinger. La oss tenke dette gjennom. Hver nabo-kvadrant i teppet er et produkt av sjanse, ifølge materialistiske ressurser. Har en svart nabo-kvadrant kjennskap til at den passer fint inn i en komplett dyne etter et geometrisk mønster av svarte, røde og gule firkanter? Med mindre hver nabo-kvadrant har en umiddelbar funksjon, vil evolusjonen ikke bevare den. Tilsvarende vil ingen folding bli "beholdt" med noe fremtidig håp om at den kan ha "verktøy for å løse det vanskelige proteinfoldeproblemet." Foldonet kunne ikke bryr seg mindre! Det måtte fungere riktig når det oppsto.
En intelligent designer kunne planlegge foldoner som en nyttig strategi for å konstruere ulike komplekse proteiner på en modulær måte. En designer kan til og med bevare nyttige foldoner, akkurat som en dataprogrammerer skriver subrutiner til bruk i andre programmer. Med mindre hver delrutine faktisk gjør noe nyttig for systemet som helhet dog, hvilken nytte er det i det? Si at du har en subrutine som sier "Gjenta hvilket som helst argument som kommer inn i inntaksregisteret." Med mindre systemet trenger den funksjonen som en del av hva den gjør, kan du kjøre underrutinen til 'kuene kommer hjem', og ingenting godt kommer av det.
Bilde 4. Proteinfolding skjer på 4-dimensjonalt nivå
 Kort sagt, reduserer ikke foldon-strategien sannsynligheten for suksess, og det løser ikke "det vanskelige proteinfoldeproblemet" for evolusjonisten. Det er bare splitt og ikke hersk.
Kort sagt, reduserer ikke foldon-strategien sannsynligheten for suksess, og det løser ikke "det vanskelige proteinfoldeproblemet" for evolusjonisten. Det er bare splitt og ikke hersk.
Englander og Mayne gjør en stor sak av "repeterende proteiner" som utgjør omtrent 5 % av det globale proteomet. Disse repeterende proteinene "har en nonglobulær kroppsplan laget av små gjentatte motiver i 20-40 overskudd-området som er samlet i et lineært utvalg." Er de gode kandidater for foldoner? Vi vet at mange proteiner inneholder repeterende strukturer som alfa-spoler og beta-svøp (sheets), men essensen av et funksjonelt protein er ikke dets repeterende deler, men deres aperiodiske deler. Vi har sett dette kravet i andre typer intelligent design, for eksempel språk. Sikker; Noen ganger utgjør en rekke streker et fint skille mellom avsnitt, men du vil ikke få mye mening ut av alle de repeterende sekvenser.
Bilde 5. Alpha-spoler og Beta-svøp
La oss se om de kan gjøre det:
"De forskjellige familier av repeterende proteiner er svært forskjellige i detaljert struktur, men innen hver familie er gjentakelsene topologisk nesten identiske. Disse observasjonene antyder at repeterende proteiner oppstod ved gjentatt duplisering på et tidlig stadium i utviklingen av større proteiner fra mindre fragmenter. Tilgjengelige eksempler viser at den globale organiseringen kan oppstå fra fortsatt repeterende vekst som lukker den lineære geometrien, og ved sammensmelting av ikke-identiske enheter, og det vil dermed videreføre deres foldon-lignende egenskaper.
Bruken av foldoner for effektiv folding av proteiner kan ses som en dominerende årsak til utvikling og oppbevaring av en foldonbasert kroppsplan gjennom proteinutvikling. I denne oppfatningen kom samtidige proteiner så konsekvent til deres modulære foldon-baserte design og deres foldbaserte foldingstrategi fordi disse sammenhengende egenskaper ko-evoluerte. Men det faktum at mange kjente foldoner bringer sammen sekvensielt fjerntliggende segmenter krever i det minste en ekstra mekanisme."
Dette høres ut som den evolusjonære historien som dupliserte gener, som ble frø til nye gener. Så hvis vi dupliserer linjen med bindestreker, og deretter bytter noen av bindestrekene til komma, vil vi komme et sted? Neppe. Hvis vi kutter ut "kan" og "kanskje" av historien, er det ikke mye igjen, uten konklusjonen om at "en ekstra mekanisme" er nødvendig for å få foldet proteiner. (Vi har en: Intelligens) Og få tak i dette: Selv om du får et polypeptid å brette seg inn til en kule, er det søppel hvis det ikke faktisk utfører en funksjon.
Når vitenskapelige materialister begynte å forholde seg til proteinfoldings-problemet, forventer de at 'partisk' energilandskap som fører til deterministiske folder, snart ville bli oppdaget. Det skjedde ikke.
"Men hvordan denne tilbøyelighet kan være kodet i den fysiske kjeden av proteinstruktur er aldri blitt oppdaget. Man antyder ganske enkelt det generelle forslaget om at det er kodet i form av landskapet og til et ad hoc-prinsipp, kalt 'minimal frustrasjon' pålagt av naturlig evolusjon".
Her angir de Axes søke-utfordring i sine egne ord:
"Kvantitativ evaluering beskrevet ovenfor viser at individuelle gjenværende overskudds-interaksjonsenergier er utilstrekkelige for å velge innfødte interaksjoner i konkurranse med det store antallet konkurrerende ikke-innfødte (nonnative) alternativer. Påstanden om at den nødvendige grad av energisk avvik er gitt av formen på et ubestemt energilandskap fordi naturen har gjort det, er - tydelig sagt - ikke en nyttig fysisk-kjemisk forklaring."
Det foldon forslaget som Englander og Mayne foretrekker, er imidlertid ikke noe bedre, til tross for deres ros av det:
"Spørsmålet er hva slags tilpasnings (conformational) søking som kan forklare prosessene og banene som frakter ufoldede proteiner til deres endelige tilstand. Den foldonavhengige definerte bane-modellen svarer direkte på hver av disse utfordringene."
 Alt de har gjort, er imidlertid å flytte utfordringene fra aminosyresekvenser til foldesekvenser. Siden foldene er sammensatt av aminosyresekvenser, er imidlertid ingenting løst; Det er fortsatt radikalt usannsynlig å komme frem til en sekvens som vil produsere et funksjonelt protein uten design. Ingen mengde evolusjonerende hånd-veiving endrer det:
Alt de har gjort, er imidlertid å flytte utfordringene fra aminosyresekvenser til foldesekvenser. Siden foldene er sammensatt av aminosyresekvenser, er imidlertid ingenting løst; Det er fortsatt radikalt usannsynlig å komme frem til en sekvens som vil produsere et funksjonelt protein uten design. Ingen mengde evolusjonerende hånd-veiving endrer det:
"Evolusjonære betraktninger knytter troverdig sammen den tidlige ko-utviklingen av foldon-baserte likevektsstruktur og foldon-basert kinetisk folding."
Så mye for empirisk fasthet. Evolusjonen gjorde det. Problemet løst.
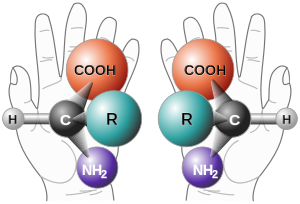
Bilde 6. Aminosyrer på V og H-form
Vi tror ikke det. For å gni det inn, vurder at Axes beregning av 1: (10^74) sekvenser er funksjonelle, er altfor generøs. Hvis vi krever at aminosyrene er venstredreiende, og krever at alle bindinger er peptidbindinger, faller sannsynligheten til 1: (10^164). For en rask demonstrasjon av hvorfor dette er håp mot alt håp, se Illustra Media's smarte animasjon fra deres film 'Origin', med tittelen "The Amoeba's Journey"her.
Oversatt av Asbjørn E. Lund (Kun Bilde 1 sto i opprinnelig artikkel. De andre er satt inn av undertegnede, se lenke i Bilde-nr.)
 biologi bekrefter vår intuisjon som livet er utformet (Harper One, 2016) -lenke.
biologi bekrefter vår intuisjon som livet er utformet (Harper One, 2016) -lenke.
 De gir navnet "foldon" til en liten kjede av aminosyrer "kanskje 15 til 35 i antall" som folder et lite stykke. Hvis polypeptidet er sammensatt av et antall av disse prefabrikerte foldon's, vil hele proteinet kanskje finne sin opprinnelige fold raskt og synke i trakten på en trinnvis måte. Eksperimenter som utfolder og refolder noen proteiner, viser faktisk denne typen trinnvis energilandskap. De liker at:
De gir navnet "foldon" til en liten kjede av aminosyrer "kanskje 15 til 35 i antall" som folder et lite stykke. Hvis polypeptidet er sammensatt av et antall av disse prefabrikerte foldon's, vil hele proteinet kanskje finne sin opprinnelige fold raskt og synke i trakten på en trinnvis måte. Eksperimenter som utfolder og refolder noen proteiner, viser faktisk denne typen trinnvis energilandskap. De liker at: Kort sagt, reduserer ikke foldon-strategien sannsynligheten for suksess, og det løser ikke "det vanskelige proteinfoldeproblemet" for evolusjonisten. Det er bare splitt og ikke hersk.
Kort sagt, reduserer ikke foldon-strategien sannsynligheten for suksess, og det løser ikke "det vanskelige proteinfoldeproblemet" for evolusjonisten. Det er bare splitt og ikke hersk. Alt de har gjort, er imidlertid å flytte utfordringene fra aminosyresekvenser til foldesekvenser. Siden foldene er sammensatt av aminosyresekvenser, er imidlertid ingenting løst; Det er fortsatt radikalt usannsynlig å komme frem til en sekvens som vil produsere et funksjonelt protein uten design. Ingen mengde evolusjonerende hånd-veiving endrer det:
Alt de har gjort, er imidlertid å flytte utfordringene fra aminosyresekvenser til foldesekvenser. Siden foldene er sammensatt av aminosyresekvenser, er imidlertid ingenting løst; Det er fortsatt radikalt usannsynlig å komme frem til en sekvens som vil produsere et funksjonelt protein uten design. Ingen mengde evolusjonerende hånd-veiving endrer det: