Om hvaler og stamfedre
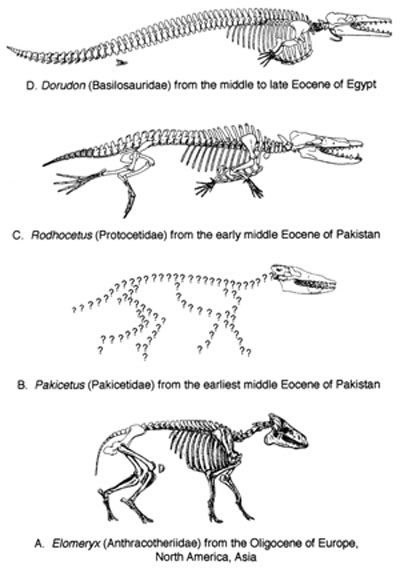 TAK: Det er merkelig at Tveter ikke engang nevner disse fossilene når han har lest om hvalens utvikling. I tillegg kan vi i dag sammenligne DNA fra hvaler med landpattedyr og andre dyr. Det er ingen tvil: hvaler stammer fra pattedyr på land.
TAK: Det er merkelig at Tveter ikke engang nevner disse fossilene når han har lest om hvalens utvikling. I tillegg kan vi i dag sammenligne DNA fra hvaler med landpattedyr og andre dyr. Det er ingen tvil: hvaler stammer fra pattedyr på land.
Bilde 1. Formodentlige forløpere til hvaler
Bilde 2. Likheten er jo 'åpenbar'
AEL: Det er ofte konflikt mellom fylogeneser basert på DNA i forhold til anatomiske baserte fylogeneser. For eks. for hvaler, som i 1960-årene ble påstått å stamme fra Mesonychider, en utdødd gruppe kjøttetende hovdyr. Men i 1990-årene viste molekylære fylogeneser større likhet til flodhest. I 2007 pekte J.G.M Thewissen og kolleger på at siden hvaler dukker opp i fossil-materialet 35 millioner år tidligere enn flodhesten, er det ikke sannsynlig at de to er nær relatert. I 2007 analyserte britiske forskere 181 molekylære og 49 morfologiske trær, og konkluderte med molekylære og morfologiske fylogeneser ofte er i strid med hverandre. Eks. på spesielle forhold: i) setefødsel -ellers ville ungen druknet ii) hvalen har ikke forbindelse mellom åndedretts-organer og munnhulen: Det gjør at den kan dukke med vidåpen munn, og få i seg masse mat uten å få det i luftrøret.. iii) Hvaler har spesielle organer for dypvanns-dykking: Hvalen klarer å lokalisere byttet nøyaktig ved hjelp av sitt unike sonarsystem. For hvalen kommer 9% av oksygenet fra lungene under dykking, mens 41% kommer fra blodet og 50% fra muskler og vev. (Tilsvarende tall for mennesket: 34% fra lungene, 41% fra blodet og 25% fra muskler/vev). iv) Hvaler har egen øre-konstruksjon: De indre knoklene i øret, har en helt annen form en hos oss. v) Hvaler har en særegen filtrerings-mekanisme: Grønnlandshvalen har ca. 350 barder med lengde opptil 4,5 m. En hval kan høste et tonn krill, fra 10.000 m3 havvann.vi) Noen hvaler kan 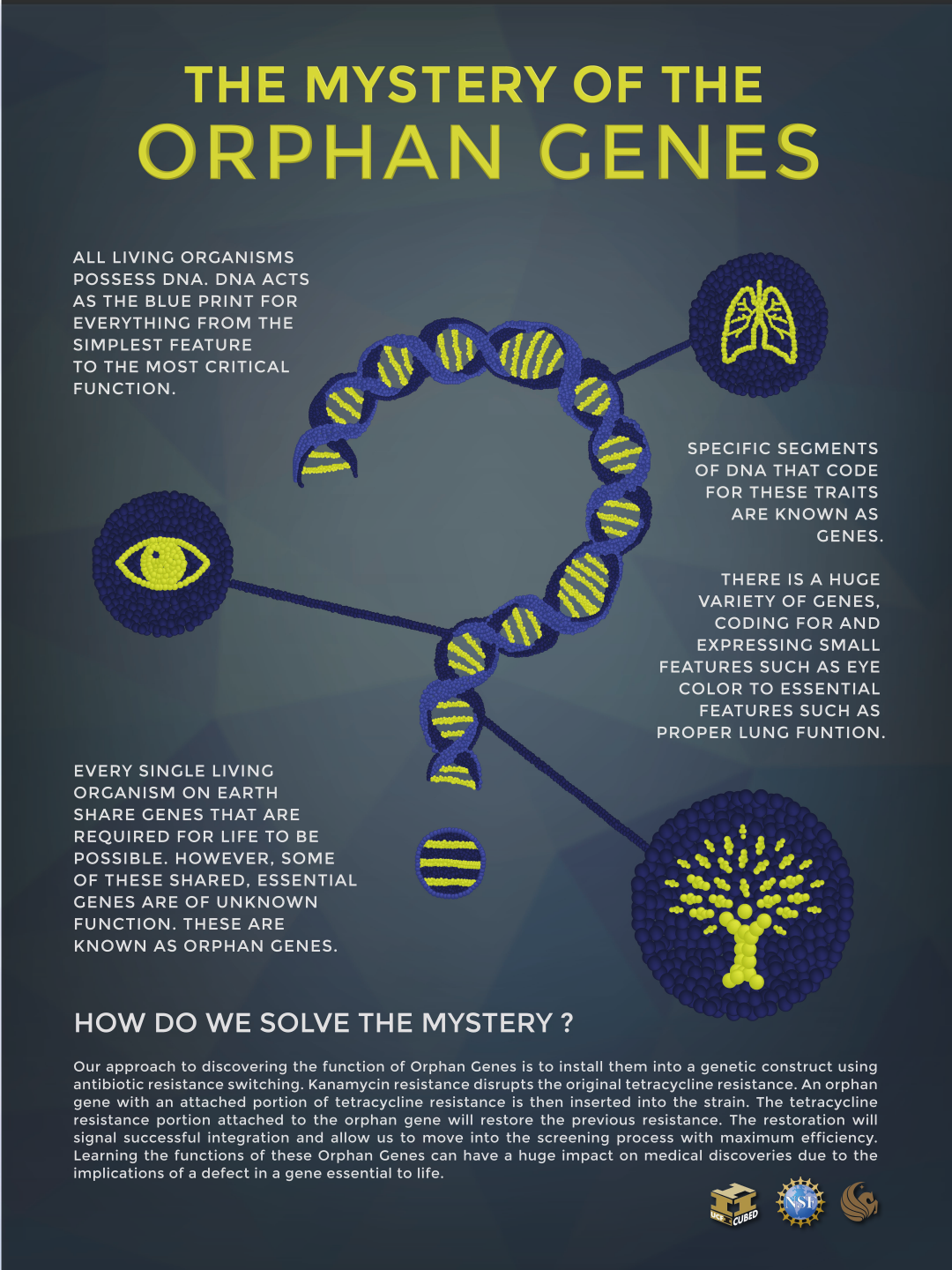 komponere musikk
komponere musikk
Forskjellen til den opprinnelige utdødde gruppen kjøttetende hovdyr(Mesonychider) kan knapt være større. Se mer om særtrekk ved hvaler her.
Bilde 3. Mysteriet orfan-gener
TAK: Det som derimot hevdes er at mennesker har en felles stamfar med sjimpansene.
AEL: Så spørs det bare om det holder.
Det som er poenget med Tveters argumentasjon, er bl.a. at: Hvert sekvensert plante, dyr og mikrobe -genom inneholder en stor del gener (ca.10-30%) som er unike for den taksonomiske gruppen. De er blitt identifisert på det viset at de mangler tilsvarende motparter i andre organismer. Et annet interessant trekk er at majoriteten av disse gruppe-spesifikke (orfan) genene også synes å være relatert til tilpasningsdyktige funksjoner, som viser ytterligere spesifisitet relatert til en bestemt gruppe organismer, noe som gjør det mulig for den å tilpasse seg og fylle økologiske nisjer. (1-3) Det er nå velkjent blant molekylære biologer at gener fungerer i integrerte, komplekse, overlappende, interavhengige og utsøkt kontrollerte nettverk. Videre er disse gen-nettverkene dynamiske og svarer til all slags stikkord og signaler, som konstant kontrolleres av celle-systemet. Det finnes ikke motstykke til dette kompleksitets- og effektivitets-nivået i systemer som er planlagt og designet mennesker. Biologer kan ta lærdom av gode programmerere. Gode programmerere vil gjenbruke deler av sin kode og syntaks, i ulike deler av sitt arbeid og også mellom ulike software prosjekter. At duplisering av ulike sekvenser dukker opp i ulike gener, tilsvarer et standard prinsipp i programmering.
Se mer på spesifikke menneskelige gener her.
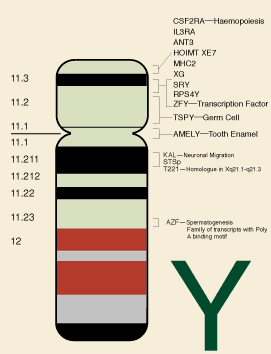
En av de mest dogme-ødeleggende rapporter om menneske-sjimpanse likhet, er den nylige publikasjonen om Y-kromosom sammenligning mellom mennesker og sjimpanser-spesielt den mannlig spesifikke regionen, som kalles MSY (4). Faktisk var ulikhetene mellom menneskets og sjimpansens Y kromosom så store at forfatterne til forskningsartikkelen kom med følgende utsagn: "Forskjellen i MSY gen-innhold hos sjimpanse og mennesket er mer sammenlignbart med ulikheten i 'autosomalt' gen-innhold i menneske og kylling, etter 310 millioners års separasjon.
Se mer om Menneske-sjimpanse likhet i kromosom Y her.
 I en oversikt over studier som sammenlignet menneske og ape -like RNAs, så erklærte forfatterne at det er identifisert et stort antall av nye ncRNA-avskriftskoder, hvorav mange av dem hadde menneske-spesifikke uttrykk (5). I en nylig studie av mennesker, sjimpanser og makaker, evaluerte forskerne 1.898 ulike linkRNA-gener i ni ulik vevstyper. 80% av mennesklig linkRNA hadde tilsvarende motparter uttrykt i sjimpanse, og bare 63% i makaker(6). Som i mange andre studier av underklasser av lncRNA, så er disse typene betydelig mer arts (taksonomisk) begrenset enn proteinkodende eksoner, og viser seg å være nøkkelegenskaper i genomet til å markere genetiske grenser mellom hva kreasjonister ville kalle 'skapte slag'. En nylig studie sammenlignet geners nabolag mellom ulike grupper som mennesker, sjimpanser, mus, rotter, kylling, sebrafisk og bananfluer (7). Det var større ulikhet mellom ulike dyregrupper i sammenlignbare gen-naboområder, enn for genomet generelt. Det gjaldt særlig gen-rekkefølge mellom to ulike organismer, og tar også i betraktning nærvær/fravær av gener når to ulike organismer blir sammenliknet. Forfatteren av dette studiet erklærte: "Overraskende nok er genene som finnes i funksjonelle naboområder og delt av ulike organismer, ikke nødvendigvis lik hverandre (orthologous). Når to arter deler funksjonelle naboområder, så kan genene som former disse, være ulike i hver art (7).
I en oversikt over studier som sammenlignet menneske og ape -like RNAs, så erklærte forfatterne at det er identifisert et stort antall av nye ncRNA-avskriftskoder, hvorav mange av dem hadde menneske-spesifikke uttrykk (5). I en nylig studie av mennesker, sjimpanser og makaker, evaluerte forskerne 1.898 ulike linkRNA-gener i ni ulik vevstyper. 80% av mennesklig linkRNA hadde tilsvarende motparter uttrykt i sjimpanse, og bare 63% i makaker(6). Som i mange andre studier av underklasser av lncRNA, så er disse typene betydelig mer arts (taksonomisk) begrenset enn proteinkodende eksoner, og viser seg å være nøkkelegenskaper i genomet til å markere genetiske grenser mellom hva kreasjonister ville kalle 'skapte slag'. En nylig studie sammenlignet geners nabolag mellom ulike grupper som mennesker, sjimpanser, mus, rotter, kylling, sebrafisk og bananfluer (7). Det var større ulikhet mellom ulike dyregrupper i sammenlignbare gen-naboområder, enn for genomet generelt. Det gjaldt særlig gen-rekkefølge mellom to ulike organismer, og tar også i betraktning nærvær/fravær av gener når to ulike organismer blir sammenliknet. Forfatteren av dette studiet erklærte: "Overraskende nok er genene som finnes i funksjonelle naboområder og delt av ulike organismer, ikke nødvendigvis lik hverandre (orthologous). Når to arter deler funksjonelle naboområder, så kan genene som former disse, være ulike i hver art (7).
Bilde 4. Y-kromosom hos mennesket
Se mer om ulikheter i gen-funksjoner her.
En av de nyeste menneske-sjimpanse DNA-metylerings undersøkelsene var den mest interessante, fordi resultatet av forskningen var totalt motsatt av det forskerne forventet. I dette prosjektet undersøkte forskerne cytosin metylerings-mønstre i genomet til blodceller fra mennesker, sjimpanser, gorillaer og orangutanger (8). De fokuserte spesifikt på 16 områder i kromosom 21 og 22, som er kjent for å være de mest like mellom menneske og ape-genom. Disse er ca 76% og 78% like respektivt (9). Segmenter av kromosomet som var for ulike, kunne ikke sammenlignes. Mønstrene og nivåene var ikke det en kunne forvente ut fra evolusjonære forutsetninger. Etter å ha undersøkt metyleringsmønstre mellom 16 regioner mellom mennesker og tre ulike aper, så benyttet de dataene til å danne et evolusjonært tre, og det var helt snudd opp-ned. Orangutanger hadde faktisk flest metylerings mønstre lik mennesket i forhold til sjimpanser og gorillaer. Dernest kom gorilla, og sjimpansen færrest. I følge standard evolusjonære mønstre, skulle det vært omvendt
Se mer om: Ulikheter menneske-sjimpanse -knyttet til epigenomet her.
 Framover: Det er tydelig at vitenskapelige rapporter ar bitt vridd til å promotere og rettferdiggjøre en sekulær agenda med degradering av menneskeverdet. De a-moralske følgende av at mennesket ikke anses som noe annet enn et dyr, er tallrike.
Framover: Det er tydelig at vitenskapelige rapporter ar bitt vridd til å promotere og rettferdiggjøre en sekulær agenda med degradering av menneskeverdet. De a-moralske følgende av at mennesket ikke anses som noe annet enn et dyr, er tallrike.
Ved å avhumanisere oss er mange smakløse oppfinnelser, som sikter på samfunnsmessig kontroll av populasjonen, rettferdiggjort. Derved kan tvilsomme tiltak, styrt av mennesker med et evolusjonært forbedrings-tankemønster, bli satt ut i livet. En bok som tar for seg dette emnet er: 'Scientific Technocracy', som viser hvordan slike dehumaniserende tiltak øker verden over (10). Det er et ideologisk underbruk av økende sentralisert, global styring, som i mange tilfeller har vist seg korrupt og ondskapsfullt . Dette er forhold som gjør emnet med menneskelig opprinnelse så viktige å forstå. Det har vært en del av grunnlaget for slike tiltak, at mennesket deler felles opphav med sjimpansen, og at vi ikke har større verdi enn andre dyr. Men som vi har vist i denne gjennomgangen, så viser hovedtyngden av vitenskapelige data at mennesker er unike, atskilt fra sjimpanse.
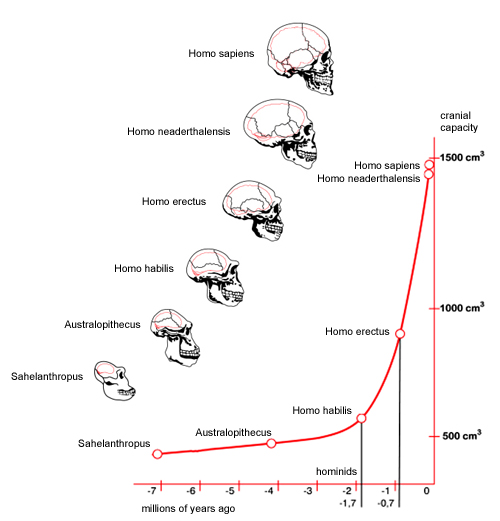
Bilde 5. For store endringer på for kort tid
Tid nok?
For proteiner: Lignende proteiner med ulike funksjoner: Lab-test: hvor mange mutasjoner trengs? Hvilke innovasjoner er mulige ut fra neo-darwinistiske mekanismer Eks. To tredimensjonale proteiner: Kbl og BioF: To lignende bakterielle proteiner, med ulike funksjoner Funksjonslikhet krevde minst 7 mutasjoner: Ikke tid nok! 3 koordinerte mutasjoner er krevende også for bakterier Beregnet ventetid for 7 mutasjoner i bakteriestamme: (10 opphøyd i 27) år Universets alder i størrelsesorden: (10 opphøyd i 10) år Konklusjon: Det å endre proteiners virkemåte er godt utenfor rekkevidde for neo-darwinistiske prosesser
For mennesker: Hva med mennesket? i) Hvor mange mutasjoner kreves for å endre en australopithecus til en Homo erectus? ii) Kan neo-darwinistiske prosesser klare det på aktuelle tiden (ca. 1.5 mill. år)? i) Bamble og Liebermann* talte 16 trekk som først dukket opp ved homo erectus Hver av dem ville kreve flere mutasjoner Det ville ta 6 millioner år for én mutasjon å bli permanent i en avstamningslinje To mutasjoner anslo de ville kreve 216 mill. år Aktuell tid tilgjengelig er 1,5 mill. år Eks. på nødvendige endringer: Stabilisere hodet, oppreist gange, lengre bein, kortere armer, finmotorikk tunge/lepper, større hodeskaller, abstrakt tenkeevne, endring i muskelstyrke.. Konklusjon: Neo-darwinistiske mekanismer utilstrekkelige
Se mer om hominider og mennesker: Del I og Del II
Referanser:
1. Tautz D. og T. Domazet-Loso. 2011. The evolutionary origin of orphan genes. Nature Review Genetics 12:692-702
2. Tomkins J. og J.Bergman. 2013. Incomplete Lineage Sorting and other 'rogue' data fell the tree of life. Journal of Creation 27:84-92
3. Yang L. et al. 2013, Genome-wide identifcations, characterization and other expression alaylysis of lineage-specific genes within zebrafish. BMC Genomics 14:65
4. Hughes J.F et al. 2010. Chimpanzee and human Y chromosomes ar remarkably divergent in structure and genen content. Nature 463: 536-539
5. Somel et al. 2013. Human brain evolution: transcripts, metabolics and their regulators. Nature Reviews Neuroscience 14:112-127
6. Washietl, S. et al. 2014. Evolutionary dynamics and tissue specificity of human long noncoding RNAs in six mammals. Genome Research Koi:10.1101/gr.165035.113
7. Al-Shahrour, F.et al. 2010. Selection upon genome archtecture: conservation of functional neighborhoods with changing genes. PLoS computational Biology. 6:e1000953
8. Fukuda et al. 2013. Regional DNA methylation differences between humans and chimpanzees are associated with genetic changes, transcriptional divergence and disease genes. Journal of Human Genetics, 58 (7): 446-454
9. Tomkins, J. S+013. Comprehensive Analysis of Chimpanzee and Human Chromosomes Reveals Average DNA Similarity of 70%. Answers Research Journal. 6 (1): 63-69
10. Colins, D.C. and P.D.Collins. 2006. The Ascendancy of the Scientific Dictatorship: An examination of Epistemic Autocracy From the 19th to the 20th Century. BookSurge LLC, www.booksurge.com
Oversettelse og utvalg av tekst og bilder ved Asbjørn E. Lund
 TAK: Det er merkelig at Tveter ikke engang nevner disse fossilene når han har lest om hvalens utvikling. I tillegg kan vi i dag sammenligne DNA fra hvaler med landpattedyr og andre dyr. Det er ingen tvil: hvaler stammer fra pattedyr på land.
TAK: Det er merkelig at Tveter ikke engang nevner disse fossilene når han har lest om hvalens utvikling. I tillegg kan vi i dag sammenligne DNA fra hvaler med landpattedyr og andre dyr. Det er ingen tvil: hvaler stammer fra pattedyr på land.
 TAK: Det er merkelig at Tveter ikke engang nevner disse fossilene når han har lest om hvalens utvikling. I tillegg kan vi i dag sammenligne DNA fra hvaler med landpattedyr og andre dyr. Det er ingen tvil: hvaler stammer fra pattedyr på land.
TAK: Det er merkelig at Tveter ikke engang nevner disse fossilene når han har lest om hvalens utvikling. I tillegg kan vi i dag sammenligne DNA fra hvaler med landpattedyr og andre dyr. Det er ingen tvil: hvaler stammer fra pattedyr på land.
 komponere musikk
komponere musikk  I en oversikt over studier som sammenlignet menneske og ape -like RNAs, så erklærte forfatterne at det er identifisert et stort antall av nye ncRNA-avskriftskoder, hvorav mange av dem hadde menneske-spesifikke uttrykk (5). I en nylig studie av mennesker, sjimpanser og makaker, evaluerte forskerne 1.898 ulike linkRNA-gener i ni ulik vevstyper. 80% av mennesklig linkRNA hadde tilsvarende motparter uttrykt i sjimpanse, og bare 63% i makaker(6). Som i mange andre studier av underklasser av lncRNA, så er disse typene betydelig mer arts (taksonomisk) begrenset enn proteinkodende eksoner, og viser seg å være nøkkelegenskaper i genomet til å markere genetiske grenser mellom hva kreasjonister ville kalle 'skapte slag'. En nylig studie sammenlignet geners nabolag mellom ulike grupper som mennesker, sjimpanser, mus, rotter, kylling, sebrafisk og bananfluer (7). Det var større ulikhet mellom ulike dyregrupper i sammenlignbare gen-naboområder, enn for genomet generelt. Det gjaldt særlig gen-rekkefølge mellom to ulike organismer, og tar også i betraktning nærvær/fravær av gener når to ulike organismer blir sammenliknet. Forfatteren av dette studiet erklærte: "Overraskende nok er genene som finnes i funksjonelle naboområder og delt av ulike organismer, ikke nødvendigvis lik hverandre (orthologous). Når to arter deler funksjonelle naboområder, så kan genene som former disse, være ulike i hver art (7).
I en oversikt over studier som sammenlignet menneske og ape -like RNAs, så erklærte forfatterne at det er identifisert et stort antall av nye ncRNA-avskriftskoder, hvorav mange av dem hadde menneske-spesifikke uttrykk (5). I en nylig studie av mennesker, sjimpanser og makaker, evaluerte forskerne 1.898 ulike linkRNA-gener i ni ulik vevstyper. 80% av mennesklig linkRNA hadde tilsvarende motparter uttrykt i sjimpanse, og bare 63% i makaker(6). Som i mange andre studier av underklasser av lncRNA, så er disse typene betydelig mer arts (taksonomisk) begrenset enn proteinkodende eksoner, og viser seg å være nøkkelegenskaper i genomet til å markere genetiske grenser mellom hva kreasjonister ville kalle 'skapte slag'. En nylig studie sammenlignet geners nabolag mellom ulike grupper som mennesker, sjimpanser, mus, rotter, kylling, sebrafisk og bananfluer (7). Det var større ulikhet mellom ulike dyregrupper i sammenlignbare gen-naboområder, enn for genomet generelt. Det gjaldt særlig gen-rekkefølge mellom to ulike organismer, og tar også i betraktning nærvær/fravær av gener når to ulike organismer blir sammenliknet. Forfatteren av dette studiet erklærte: "Overraskende nok er genene som finnes i funksjonelle naboområder og delt av ulike organismer, ikke nødvendigvis lik hverandre (orthologous). Når to arter deler funksjonelle naboområder, så kan genene som former disse, være ulike i hver art (7). Framover: Det er tydelig at vitenskapelige rapporter ar bitt vridd til å promotere og rettferdiggjøre en sekulær agenda med degradering av menneskeverdet. De a-moralske følgende av at mennesket ikke anses som noe annet enn et dyr, er tallrike.
Framover: Det er tydelig at vitenskapelige rapporter ar bitt vridd til å promotere og rettferdiggjøre en sekulær agenda med degradering av menneskeverdet. De a-moralske følgende av at mennesket ikke anses som noe annet enn et dyr, er tallrike.